 论文:https://www.nature.com/articles/s41597-023-02125-y
论文:https://www.nature.com/articles/s41597-023-02125-y
摘要
训练计算机视觉模型的成功在很大程度上依赖于带有注释的大规模真实图像的支持。然而,由于隐私保护和需要大量标注,在病理学中难以获得这样的标注数据集。为了辅助计算病理学,合成数据生成、管理和标注提供了一种经济有效的方法增强数据多样性,可以在不同阶段快速提高模型性能。本文提出了一个大规模的合成病理图像数据集,带有细胞核语义分割标注,称为合成细胞核和标注向导(SNOW)。本文提出的 SNOW 是通过一个标准化的工作流程,应用现成的图像生成器和核标注器来开发的。该数据集包含 20k 个图像块和 1,448,522 个标注的细胞核
本文证明了 SNOW 可以用于监督和半监督训练场景。广泛的结果表明,合成数据训练的模型在各种模型训练设置下具有竞争力,扩大了更好地使用合成图像来增强下游数据驱动的临床任务的范围
引入
全视野数字切片WSI提供了一种有效的工具来评估疾病的视觉状态,从分期、转移到预后。特别是高分辨率的WSI可以捕获不同的组织外观和细胞形态,为临床决策提供信息。为了提前诊断乳腺癌,细胞核鉴定起着关键作用,因为其特征与患者预后密切相关。越来越多的深度学习方法被开发用于自动化基于WSI的核分割,然而模型的性能取决于现实世界训练样本的质量和数量
数据集的准备和构建对于病理学中的深度学习应用变得至关重要,目前的标准是在精心设计的现实世界数据集上训练模型网络,并对模型进行微调,直到收敛
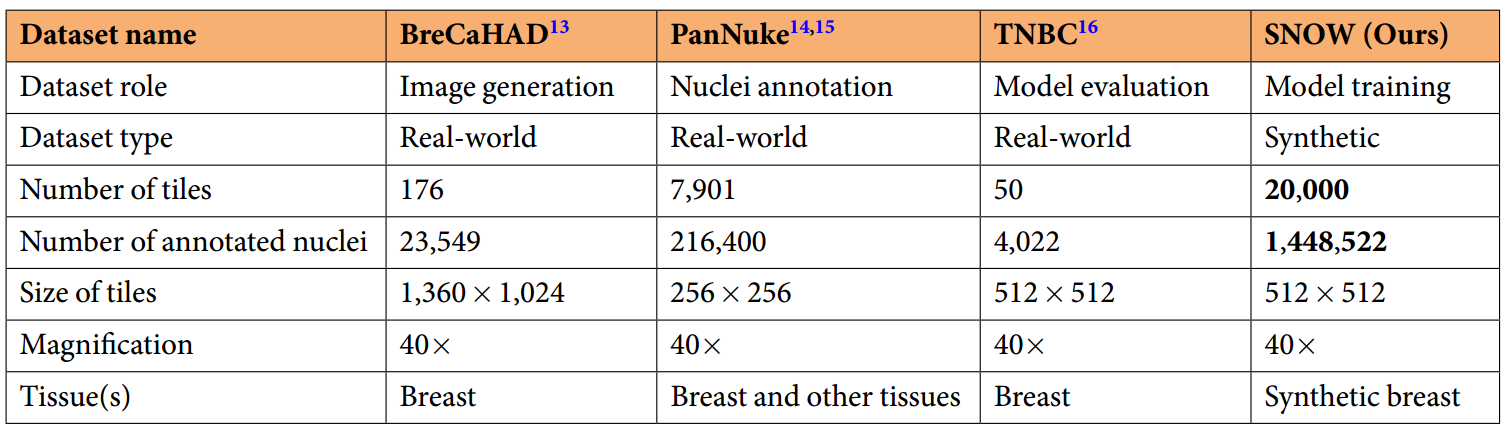
然而,由于隐私保护和需要大量的标注,病理数据管理在医疗保健系统中面临着艰巨的挑战。从复杂的WSI中标注组织特征平均需要 1.8 小时,因此人类尺度的WSI标注越来越具有挑战性。迄今为止,真实世界的乳腺癌相关细胞核数据集涵盖了有限范围的样本,来自BreCaHAD、PanNuke和TNBC,这些数据仍然不足以达到深度学习所需的规模。相反,合成数据生成提供了一种具有成本效益的方法来实现数据多样性,有望促进不同阶段下游任务的模型训练
生成对抗网络GANs已经成为生成高质量合成图像的关键计算工具,但是基于Gan的方法无法直接生成相应的核分割结果(即核掩码)。为了解决这一限制,在癌症病理学中,研究人员应用掩模定义的方法来生成合成图像,以改善细胞语义分割(即,将每个图像像素分类为背景或细胞核)。这些方法通常从提出预先定义的掩模来定位原子核位置开始。在这样的掩模约束设置下,使用Gan容易只生成形状预定义的核样本。因此,这个预定义的设置成为训练健壮模型以分割具有挑战性的单元格的障碍
为了应对缺乏大规模图像数据集的问题,本文从标准化、可扩展和可复制的角度引入了一个基于合成数据生成和注释的数据集。本文提出了合成核和标注向导(SNOW),它允许在没有任何先前掩模约束的情况下生成乳腺癌细胞核的多种合成数据。本文的主要重点放在合成数据集管理上,这与寻求模型参数优化或生成真实世界数据集的研究有很大不同
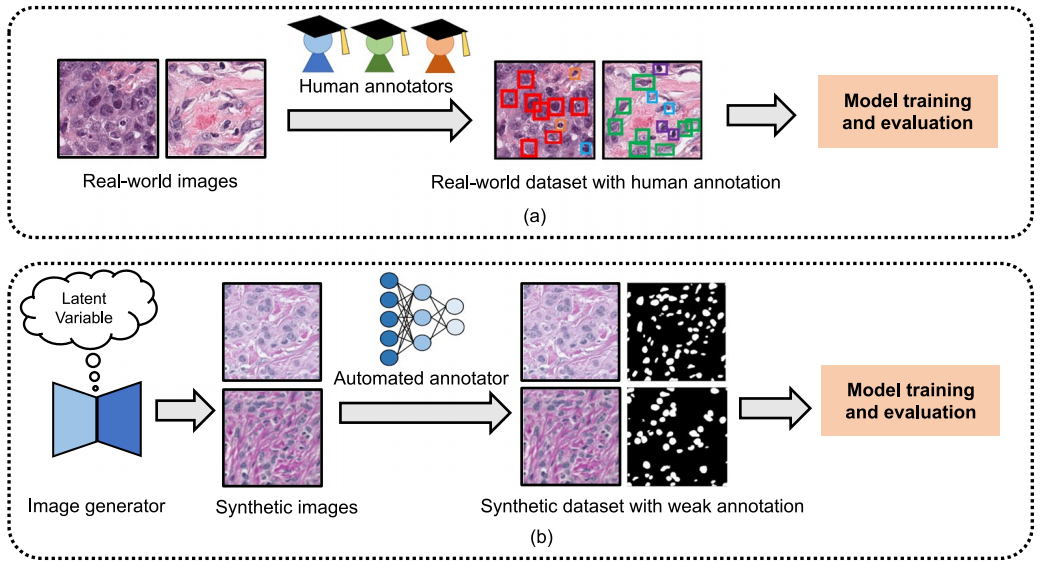
本文在研究中解决了有关合成数据集生成、评估和质量的数据中心问题。首先,能否通过使用有限的公共训练数据,以最少的人力来生成信息丰富的合成样本?第二,使用大规模合成图像进行核语义分割时,需要哪种类型的训练策略?最后,合成图像是否可以单独作为真实世界数据集的替代品来进行核语义分割?
SNOW 数据集为病理图像评估中有意义的合成数据的应用开辟了新的视角。这个简单而有用的方法,连同对图像内容的质量验证,已经证明了单独来自合成图像的训练模型可以获得具有竞争力的核语义分割性能。总之,SNOW 将成为深度学习核分割的关键资源,扩大计算病理学中数据管理的前景。此外,SNOW 还可以潜在地促进各种基于图像的下游任务,如肿瘤分期、预后和分子分析。SNOW 还可以促进乳腺癌特异性的预训练和微调任务。此外,SNOW 数据集可以作为训练对抗性攻击检测模型的源数据,用于医学图像分析中的恶意攻击识别。
方法
工作流程

SNOW 包含用于乳腺癌病理图像的合成图像生成器SIG和细胞核标注器NA。SIG根据有限的病理训练数据生成生动的数据,NA被设计生成弱细胞核标注,而不需要增加细胞核标注器的微调过程。这里的SIG和NA不是在大规模域外数据集上进行预训练,而是使用域内公共数据集从头开始训练,因为它们在图像分辨率和注释可用性方面具有差异特征。这种设计为生成成对的合成图像样本和标注提供了一种有效且可重现的方法。SNOW 在无需人工标注工作的情况下扩展了当前病理细胞核分析的数据规模
合成图像生成器 SIG
本文将合成图像生成器定义为生成合成图像块的有效工具,包括组织和细胞核。本文对合成图像生成器的设计和目的提出了两个指导原则,包括图像质量和数据生成效率
合成图像质量是通过测量合成数据与真实数据之间的相似性来评估的,包括细胞核形状和组织形态。此外,每幅合成图像中的细胞核数量等核统计信息也反映了合成图像的质量。为了满足这些要求,选择StyleGAN2作为SIG,因为它具有很强的图像生成能力。StyleGAN2生成器将从正态分布得到的的潜在编码 z\in Z 映射到真实图像。潜在编码z首先通过映射函数映射到中间潜在编码 w\in W。然后,将w变换为k个向量作为样式信息注入到样式块中。为了确保良好的图像质量,StyleGAN2使用了感知路径长度PPL正则化,该正则化用于评估潜在空间内插的质量,并与形状的一致性和稳定性相关。此外,StyleGAN2应用截断操作以通过避免所生成的图像收敛到训练数据集的平均表示来为图像生成添加更多的多样性。这些组件使StyleGAN2能够生成任务中所需的合成图像
在病理图像分析领域,为了保证StyleGAN2生成图像的质量,数据生成效率是一个至关重要的因素。考虑到小规模的训练数据集,StyleGAN2的鉴别器可能会过度拟合训练样本,其中对StyleGAN2生成器的反馈变得没有意义,并且训练开始偏离。标准数据增强有助于缓解过度拟合,但必须很好地处理噪声增强输入。为了解决这些问题,本文在SIG中使用了自适应鉴别器增强ADA方案,而不改变StyleGAN2的损失函数或网络结构
Ada方案通过基于潜在过适应程度动态地使用阈值 p\in [0,1] 来控制增强强度。以概率 p 应用增强或以概率 1-p 跳过)。在RandAugment的启发下,增强中包括 18 个不同的转换。该算法通过评估原始训练样本上的StyleGan2的输出与生成的图像之间的相关性来调整截止点p。此外,ADA通过估计训练样本的部分来评估过拟合度,以获得正的鉴别器输出。在没有大规模域外训练的情况下,ADA的使用被证明有助于数据受限情况下的训练
细胞核标注器 NA
本文将细胞核标注器NA定义为标记效率高的专家,以自动完成合成图像中的细胞核标注。NA模块不追求细粒度的标注,而是只产生一组弱核标注,这些标注在训练分割模型时表现得相当好,而不需要增加人工校正。目前,细胞核语义分割的常规工作流程严重依赖于人工注释,这解释了文献中细胞核数据集规模的限制。与使用人类标注来扩大数据集相反,本文通过利用关于细胞核输入的人类先验知识的公共数据集来初始化NA模块
本文使用HoVer-Net作为细胞核标注器,因为它在分割具有挑战性的细胞核示例的多个基线中具有可靠的性能。在 HoVer-Net中,特征提取组件的灵感来自于 50 层的预激活残差网络Preact-ResNet50。与标准Preact-ResNet50相比,HoVer-Net通过在第一个卷积中使用 1 的步长并删除后续的maxpooling操作,将总下采样因子从 32 减少到 8。这些修改减少了信息的直接丢失并确保了分割的质量。HoVer-Net通过不同的功能分支(包括核像素(NP)和HoVer分支)利用最近邻上采样。 NP分支预测像素与核或背景的隶属关系,而HoVer分支预测核像素与其质心的水平和垂直距离
训练策略
数据
由于具有组织的高分辨率详细视图(1360*1024),本文使用BreCaHAD来确保合成图像生成器的训练性能。 BreCaHAD数据集包括 162 个乳腺组织图块,其中包含每个细胞核的点位置。由于BreCaHAD数据集不提供整个细胞核的语义分割掩模(例如,仅点注释),因此利用PanNuke数据集来训练细胞核标注器,其中包含图像和相应的注释掩模。迄今为止,PanNuke数据集是最大的细胞核分割数据集。根据拟议的流程,SNOW 产生两万个合成乳腺癌组织 patch,并配有细胞核形状标注
技术验证
实验设置
应用细胞核语义分割来评估所提出的合成数据集的性能。为了严格避免数据混乱,本文在分割模型的训练过程中没有使用额外的数据增强。本文的主要重点是诊断数据效用,这与大多数调整模型参数以优化分割性能的研究不同。在实验中,本文考虑标准的ResNet34、DenseNet和Xception作为UNet架构中的编码器,它们广泛应用于医学图像分割。模型训练后,评估了三阴性乳腺癌 (TNBC) 数据集 16 的独立真实数据集上的分割性能。对于本文研究中的所有实验,批量大小为 64,优化器为Adam,学习率为 1e-4。本文使用四个 Tesla V100 SXM2 GPU 进行实验。为了评估核语义分割性能,本文使用 DICE 分数、Jaccard 指数(也称为 Intersection-OverUnion (IoU))和平均 hausdorff 距离 (aHD)
对于合成图像生成器,本文使用BreCaHAD训练的StyleGAN2-ADA模型。模型权重是通过在BreCaHAD数据集上从头开始训练StyleGAN2-ADA获得的。批量大小为 64,优化器为Adam,学习率为 2.5e-3。 BreCaHAD的每张图像都被裁剪成分辨率为 512×512 的 patch 用于模型训练。选择BreCaHAD作为生成器输入的选择,因为它主要关注核评估,其中每个图像块都包含合理数量的核用于模型训练。这与癌症基因组图谱 (TCGA) 数据库形成鲜明对比,该数据库包含未选择和注释细胞核、基质细胞、淋巴细胞和其他组织内容的WSI。对于图像标注器的设计,使用PanNuke训练的HoVerNet模型。在训练过程中,batch size为 8,优化器为Adam,初始学习率为 1e-4,25 个 epoch 后降至 1e-5
